Beispiel-Workflow: Ausführung von EO Processing MPI-Jobs auf einem SLURM-Cluster auf CODE-DE FRA1-1
Das Ziel dieses Artikels ist es, die Ausführung von MPI-Jobs auf einem SLURM-Cluster auf FRA1-1 zu zeigen.
MPI - Message Passing Interface - ist der Standard für die Kommunikation zwischen Rechnerknoten in einer parallelen Rechnerarchitektur. Ein Programm, das unter Verwendung einer MPI-Implementierung geschrieben wurde, ermöglicht es, jeden Knoten mit der Ausführung eines bestimmten Teils der verteilten Aufgabe zu beauftragen. Die Knoten können sich dann gegenseitig über den Status ihrer spezifischen Aufgaben informieren, so dass z. B. ein Knoten dort weitermachen kann, wo ein anderer aufgehört hat. Aus der Sicht des Entwicklers wird der MPI-Code in einem zentralen Skript geschrieben, so als ob er für eine einzelne Maschine geschrieben wäre.
Was wir behandeln werden
Installieren von mpi4py – Implementierung von OpenMPI in Python
Installation von snappy – eine Python-Bibliothek für die Analyse und Verarbeitung von Satellitenbildern
Zusätzliche Python-Module installieren: s3cmd, boto3 und numpy.
Installieren Sie das EO-Datennetzwerk auf allen Knoten und fügen Sie so dem Cluster EO-Datenfunktionen hinzu.
Verteilen eines MPI-Jobs auf dem Cluster.
Ausführen dieses MPI-Jobs zur einfachen Vorverarbeitung von Satellitenbildern
Herunterladen und Anzeigen eines der im SLURM-Cluster verarbeiteten Bilder
Voraussetzungen
Nr. 1 Konto
Sie benötigen ein CODE-DE Konto mit Zugriff auf die Horizon-Schnittstelle: https://cloud.fra1-1.cloudferro.com/auth/login/?next=/.
Nr. 2 Ein laufender SLURM-Cluster
Wir verwenden das Cluster-Setup aus dem Artikel:
Beispiel eines SLURM-Clusters auf CODE-DE FRA1-1 Cloud mit ElastiCluster.
Dieser Cluster hat einen Master- und 4 Worker-Knoten, er heißt myslurmcluster und hat das /home Verzeichnis als NFS-Freigabe auf allen Knoten. Außerdem wird auf den SLURM-Knoten Ubuntu 18.04 mit Python 3.6 laufen, was ideal für SNAPPY ist.
Bitte passen Sie die folgenden Befehle und Skripte an Ihre SLURM-Installation an.
Nr. 3 Grundlegende Kenntnisse in der allgemeinen Programmierung
Erfahrung im Umgang mit Linux und mit der Programmierung in einer Sprache wie Python sowie mit Bibliotheken oder Software wie: OpenMPI, Snappy, mpi4py.
Weitere Informationen finden Sie unter diesen Links:
Nr. 4 SNAP und s3cmd installieren
Zusätzlich benötigen Sie SNAP und s3cmd, die im weiteren Verlauf des Artikels heruntergeladen und installiert werden.
Schritt 1 Installieren Sie mpi4py
OpenMPI ist einer der Standards für die MPI-Implementierung. Es ist bereits auf dem SLURM-Cluster vorinstalliert, wenn Sie die Anleitung in Voraussetzung Nr. 2 befolgt haben.
Greifen wir auf den Master-Knoten zu und stellen wir sicher, dass wir uns im Verzeichnis /home/eouser befinden:
elasticluster ssh myslurmcluster
cd /home/eouser
Beachten Sie, dass /home eine NFS-Freigabe auf dem Master-Knoten ist, die (mit Unterverzeichnissen) auch auf den Arbeiterknoten eingehängt ist. Wenn Sie also den Inhalt dieses Ordners auf dem Hauptknoten aktualisieren, ist er auch für die Arbeitsknoten verfügbar.
Führen wir die folgenden Befehle (vom Master-Knoten aus) aus, um mpi4py sowohl auf dem Master als auch auf den Workern zu installieren. Der Befehl srun mit dem Flag –nodes wird für die Installation auf den Workern verwendet:
sudo apt install python3-mpi4py
srun --nodes=4 sudo apt install python3-mpi4py
Dann erstellen wir ein kleines Programm, um die mpi4py-Installation zu überprüfen
touch mpi4py_hello.py
nano mpi4py_hello.py
mpi4py_hello.py
from mpi4py import MPI
comm = MPI.COMM_WORLD
print("Hello, my rank is: " + str(comm.rank))
Führen Sie es mit folgendem Befehl aus:
mpirun -n 4 python3 mpi4py_hello.py
Dies ist das Ergebnis: .. image:: mpi_mpi4py_ok.png
Wir sehen eine Warnung über OpenFabrics und MCA-Parameter. Für die Beispiele in diesem Artikel werden wir solche Warnungen ignorieren. Es gibt auch zwei Möglichkeiten, sie zu beseitigen; eine ist die Verwendung des Parameters
--mca btl_base_warn_component_unused 0
Der Befehl würde dann lauten
mpirun -n 4 --mca btl_base_warn_component_unused 0 python3 mpi4py_hello.py
und das Ergebnis ist viel klarer:
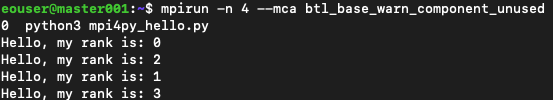
Die andere Möglichkeit, die Warnung dauerhaft zu unterdrücken, besteht darin, die Datei /etc/openmpi/openmpi-mca-params.conf zu bearbeiten und die Zeile btl_base_warn_component_unused = 0 einzufügen.
Schritt 2 snappy installieren
Snappy ist eine Python-Erweiterungsbibliothek für eine Desktop-Anwendung - SNAP, die von der ESA (European Space Agency) für die Analyse und Verarbeitung von Satellitenbildern angeboten wird.
Wir können den neuesten Link von der Download-Seite für SNAP <https://step.esa.int/main/download/snap-download/>`_ nehmen und den Download mit wget unter Verwendung des folgenden Befehls ausführen. Anschließend müssen wir diese Datei ausführbar machen:
wget https://download.esa.int/step/snap/9.0/installers/esa-snap_all_unix_9_0_0.sh
chmod +x esa-snap_all_unix_9_0_0.sh
Wir werden das Installationsprogramm im unbeaufsichtigten Modus ausführen. Auf diese Weise können wir den Prozess automatisieren und vermeiden die Fragen des interaktiven Installationsprogramms zur Konfiguration der einzelnen Knoten.
Zu diesem Zweck bereiten wir eine Antwortdatei (.varfile) vor und führen das Installationsprogramm mit der Option -q aus. Sie können die Datei nach Ihren Wünschen ändern, z. B. um andere Erweiterungen zu aktivieren.
Die .varfile sollte sich am selben Ort befinden wie die ausführbare Datei des Installationsprogramms (siehe unten).
touch esa-snap_all_unix_9_0_0.varfile
nano esa-snap_all_unix_9_0_0.varfile
esa-snap_all_unix_9_0_0.varfile
executeLauncherWithPythonAction$Boolean=true
extendPathEnvVar$Boolean=true
forcePython$Boolean=true
pythonExecutable=/usr/bin/python3.6
sys.component.3109$Boolean=true
sys.component.RSTB$Boolean=true
sys.component.S1TBX$Boolean=true
sys.component.S2TBX$Boolean=true
sys.component.S3TBX$Boolean=true
Sobald die Antwortdatei fertig ist, führen Sie das Installationsprogramm mit diesem Befehl aus:
./esa-snap_all_unix_9_0_0.sh -q
Der nächste Schritt besteht darin, snappy für unsere Python-Distribution zu aktivieren. Führen Sie die folgenden Befehle auf dem Master aus:
cd ~/snap/bin
./snappy-conf /usr/bin/python3.6
Das ist das Ergebnis:
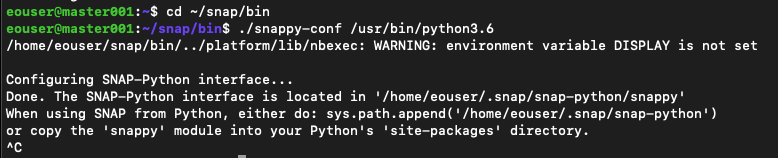
Sie können den Vorgang mit Strg+C unterbrechen, sobals Sie die Meldung „done“ sehen.
Wir werden das snappy-Modul in das Site-Packages-Verzeichnis von Python kopieren, um die Ausführung von Skripten von jedem Ort in unserem Verzeichnisbaum (sowohl auf dem Master- als auch auf den Worker-Knoten) zu ermöglichen.
sudo cp ~/.snap/snap-python/snappy /usr/lib/python3/dist-packages -r
srun --nodes=4 sudo cp ~/.snap/snap-python/snappy /usr/lib/python3/dist-packages -r
Überprüfen Sie auf dem Master-Knoten, ob es funktioniert, indem Sie die Python-Konsole aufrufen und versuchen, snappy zu importieren:
python3
>>> import snappy
Wenn alles gut geht, sollte ein Bildschirm ähnlich dem folgenden erscheinen:
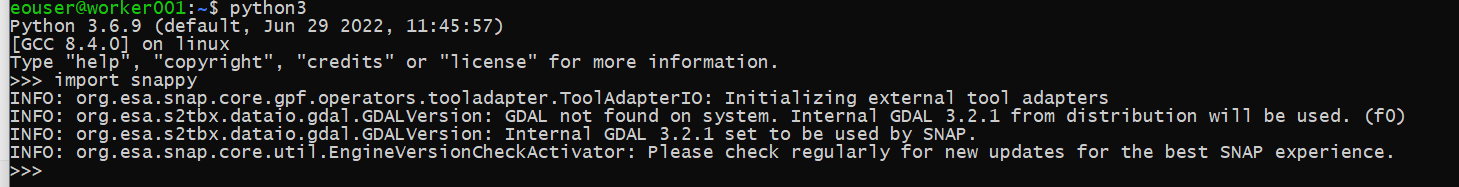
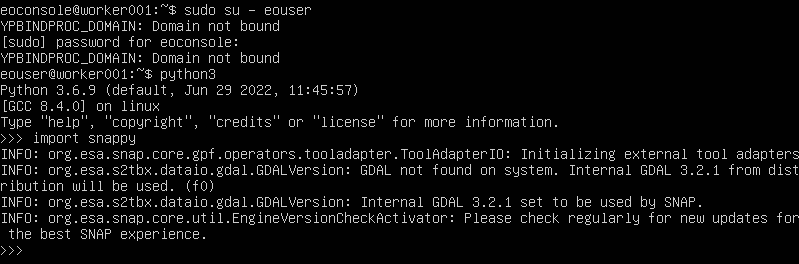
Wiederholen Sie diesen Vorgang auch auf einem der Worker Nodes. Am einfachsten ist es, die Konsole in Horizon zu verwenden; klicken Sie auf den Namen der Worker-Node-Instanz, dann auf Console, melden Sie sich als Benutzer eoconsole an, geben Sie das Passwort ein und folgen Sie den Befehlen wie im folgenden Bild:
Schritt 3 Hinzufügen von EO-Datenkapazitäten zum Cluster
Einfügen des EO-Datennetzwerks in unsere Master- und Worker-Knoten
Unser snappy-Skript wird Satellitenbilder aus dem EO-Datenspeicher abrufen. Daher sollten wir das EO-Datennetzwerk zu unseren Master- und Worker-Knoten hinzufügen.
Melden Sie sich jetzt von der Clustermaschine ab:
exit
Geben Sie die folgenden Befehle von Ihrem Arbeitsplatz aus ein, wobei Sie eodata_00341_3 durch den Namen des Ihnen zugewiesenen EO-Datennetzwerks ersetzen müssen. Um den genauen Namen dieses Netzwerks anzuzeigen, verwenden Sie die Menübefehle Netzwerk -> Netzwerke:
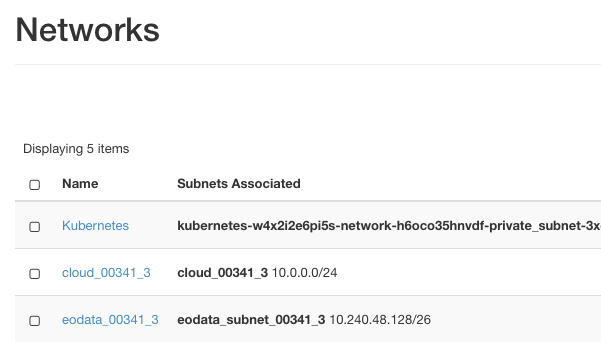
Aus dem Bild geht hervor, dass der benötigte Name eodata_00341_3 lautet. Die entsprechenden Befehle lauten:
openstack server add network myslurmcluster-master001 eodata_00341_3
for i in `seq 1 4`; do openstack server add network myslurmcluster-worker00$i eodata_00341_3; done
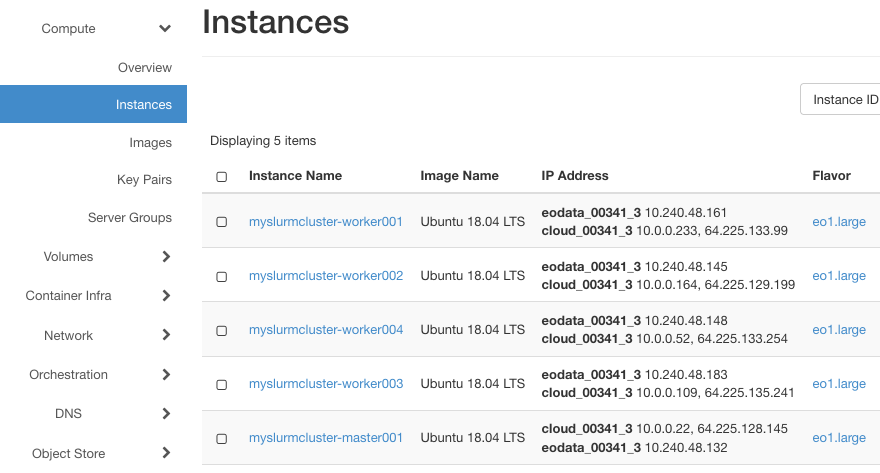
In Horizon sollten Sie sehen, dass EO-Daten erfolgreich zu den Rechnern hinzugefügt wurden:
Melden Sie sich dann mit wieder am Masterknoten an:
elasticluster ssh myslurmcluster
Installieren Sie das s3cmd-Paket
Installieren Sie das s3cmd-Paket, um SAFE-Produkte aus dem EO-Datenspeicher herunterzuladen:
sudo apt install -f s3cmd
srun --nodes=4 sudo apt install -f s3cmd
Wir müssen s3cmd für die Verwendung des EO-Datenclusters konfigurieren. Zu diesem Zweck führen Sie aus:
s3cmd --configure
und folgen Sie den Schritten des Assistenten mit diesen Werten:
access key, secret key: Geben Sie hier ihr S3-Schlüsselpaar ein
default region: Region1
S3 endpoint: eodata.cloudferro.com
Https: No
Für die anderen Werte übernehmen Sie einfach die Standardwerte (drücken Sie dazu die Eingabetaste) und speichern Sie die Konfiguration abschließend.
Schritt 4 Zusätzliche Python-Module installieren
Installieren Sie schließlich zusätzliche Python-Module für das Datenverarbeitungsskript: numpy und boto3. Sobald diese Pakete im Benutzerbereich installiert sind, werden sie auch auf den Arbeitsknoten über eine NFS-Freigabe installiert. Führen Sie zu diesem Zweck die folgenden Befehle vom Master aus aus:
python3 -m pip install boto3
python3 -m pip install numpy
Schritt 5 Ausführen eines Bildverarbeitungs-MPI-Jobs
Unser Beispielskript holt Satellitenbildprodukte im SAFE-Format aus dem EO-Datenspeicher und führt grundlegende EO-Verarbeitungsvorgänge mit SNAP/Snappy durch. Der Workflow läuft in der folgenden Reihenfolge ab:
Eine Teilmenge von EO-Datenprodukten wird nur auf einem der Knoten mit boto3 aufgelistet.
Die Liste wird in Chunks aufgeteilt, und die MPI-Funktion scatter verteilt die Chunks auf die Worker.
Jeder Worker lädt seine Teilmenge der Produkte mit s3cmd herunter.
Jeder Worker führt dann die Verarbeitung des Bildes durch. Die Wahl der snappy/SNAP Prozessoren dient nur der Veranschaulichung: Wir verwenden die SNAP-Funktion Resample und anschließend die Funktion Subset.
Nachdem das Bild verarbeitet wurde, werden die resultierenden TIFF-Dateien im gemeinsamen NFS-Ordner gespeichert.
Erstellen und bearbeiten Sie die Datei image_processing.py:
touch image_processing.py
nano image_processing.py
Geben Sie dann den folgenden Inhalt ein:
image_processing.py
import boto3
import os
import numpy as np
from mpi4py import MPI
import snappy
from snappy import ProductIO
from snappy import HashMap
from snappy import GPF
# MPI's retrieved parameters: number of all nodes on the cluster (size), and the current node (rank)
comm = MPI.COMM_WORLD
rank = comm.Get_rank()
size = comm.Get_size()
# boto3 setup: authorization and information about the subset of EO data repository to use (here Sentinel-2 L1C)
s3_resource = boto3.resource('s3', aws_access_key_id='ANYKEY', aws_secret_access_key='ANYKEY', endpoint_url='http://eodata.cloudferro.com')
s3_client = s3_resource.meta.client
bucket_name = 'DIAS'
prefix = 'Sentinel-2/MSI/L1C/2022/10/01/'
max_keys = 8
# using boto3 we generate a list of products, the list is generated only on one of the nodes (MPI's comm.rank==0)
# we split the list into chunks, then the MPI's comm.scatter function distributes the chunks between all nodes
sendbuf = []
if comm.rank == 0:
collection_dicts = s3_client.list_objects(Delimiter='/', Bucket=bucket_name, Prefix=prefix, MaxKeys=max_keys)['CommonPrefixes']
collections = np.array([i['Prefix'] for i in collection_dicts])
chunked_collections = np.array_split(collections, size)
sendbuf = chunked_collections
collections_chunk = comm.scatter(sendbuf, root=0)
# download .SAFE product files to local folder using s3cmd
for col in collections_chunk:
product_ex_prefix = col.replace(prefix, '')
cmd = 'mkdir ' + product_ex_prefix
os.system(cmd)
cmd = 's3cmd get ' + '--recursive s3://EODATA/' + col + ' ~/' + product_ex_prefix
os.system(cmd)
# Read product to SNAP and apply SNAP's Resampling
product = ProductIO.readProduct(product_ex_prefix)
paramsRes = HashMap()
paramsRes.put('targetResolution',20)
productRes = GPF.createProduct('Resample', paramsRes, product)
# Apply SNAP's Subset and save the file back to local folder
paramsSub = HashMap()
paramsSub.put('sourceBands', 'B2,B3,B4')
paramsSub.put('copyMetadata', 'true')
productSub = GPF.createProduct('Subset', paramsSub, productRes)
ProductIO.writeProduct(productSub, product_ex_prefix, 'GeoTiff')
Führen Sie das Skript mit dem folgenden Befehl aus:
mpirun --n 4 python3 image_processing.py
Nach Abschluss des Vorgangs sehen wir, dass 8 Sentinel-2 SAFE-Produkte heruntergeladen und 8 tif-Ausgabedateien erzeugt wurden.
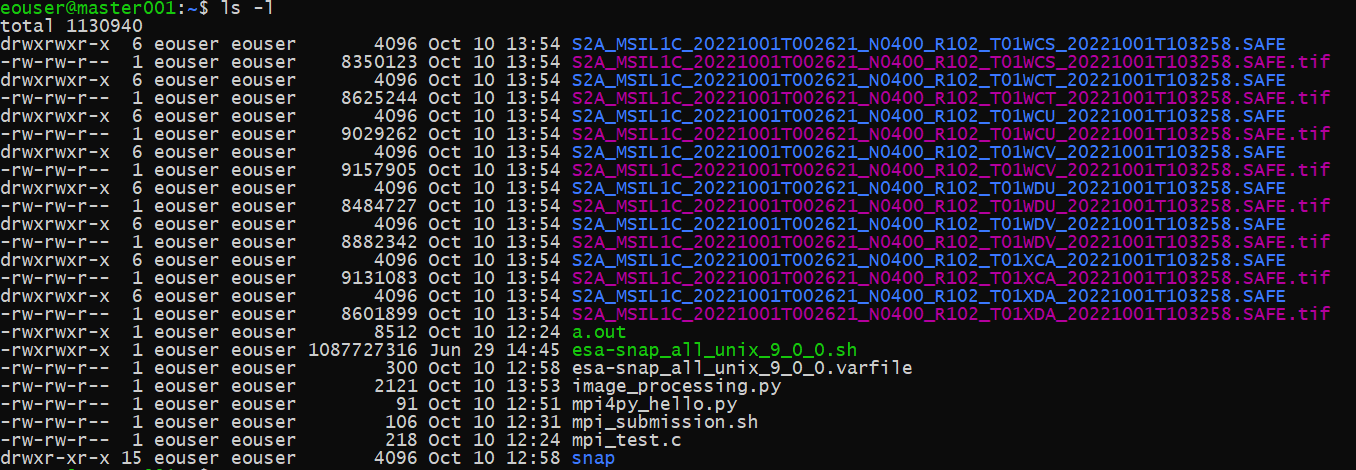
Was als nächstes zu tun ist
Wenn Sie den SLURM-Cluster für die Arbeit mit Satellitendaten verwenden, können Sie die erzeugten Bilder mit dem folgenden Befehl herunterladen
elasticluster sftp myslurmcluster
Zur visuellen Darstellung und Kontrolle der Ergebnisse, sollten Sie eine Bildverarbeitungssoftware wie SNAP-Desktop verwenden.