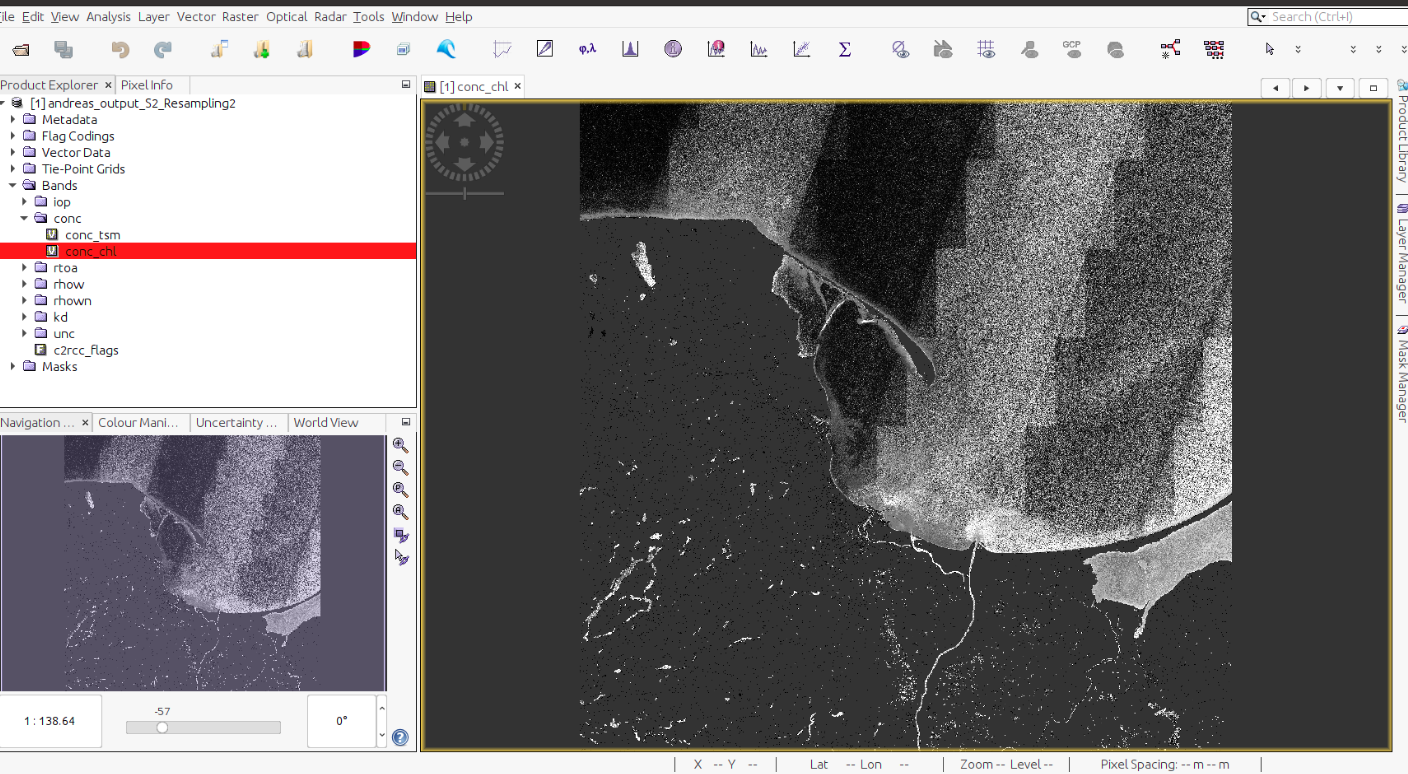
C2RCC als Beispiel für einen ESA SNAP / Snappy Prozessor auf einer CODE-DE VM
Dieses Tutorial behandelt das Starten von SNAP-Prozessoren/Operatoren über Python-Codes mit Snappy. Der Prozessor C2RCC wird als Beispiel verwendet. Mit C2RCC können Wasserinhaltsstoffe wie Schwebstoffe und Chlorophyllgehalt aus Sentinel-2 Daten abgeleitet werden. Sie finden mehr über den Prozessor selbst unter https://c2rcc.org/.
Voraussetzungen
Nr 1. Benutzerkonto
Sie benötigen ein CODE-DE-Benutzerkonto mit Zugriff auf die Horizon-Schnittstelle: https://cloud.fra1-1.cloudferro.com/auth/login/?next=/.
Nr 2. Virtuelle Maschine mit Linux Betriebsystem und Zugriffe auf das /codede Repository
Sie benötigen eine VM mit Linux Betriebsystem und Zugriffe auf das /codede Repository. In unserem Beispiel verwenden wir Ubuntu 22.04 LTS und verbinden uns mit der VM über das ssh-Protokoll (siehe auch:Wie verbindet man sich unter Linux über SSH mit einer virtuellen Maschine?)
Eine VM der Größe eo2.2xlarge ist ausreichend zur Installation von ESA SNAP (einschliesslich aller verfügbaren Werkzeuge), anspruchsvollere Verarbeitungen erfordern allerdings stärkere Varianten (vor allem einen erhöhten RAM-Bedarf).
Nr 3. Erfolgreiche Installation von ESA SNAP auf einer Linux VM
/cloud/Installation-of-ESA-SNAP-on-an-CODE-DE-VM
No 4. Erfolgreiche Kopplung von ESA SNAP mit Python auf einer Linux VM
ESA SNAP processors
Auf Ihrer Linux VM können Sie nach der erfolgreichen Installation (Nr. 3 und 4 von oben) die verfügbaren Prozessoren/Operator überprüfen, indem Sie folgenden Befehl ausführen:
/home/eouser/snap/bin/gpt -h
Anschliessend können Sie Informationen zu einzelnen Prozessoren anhand der Namen einsehen (C2RCC in diesem Beispiel):
/home/eouser/snap/bin/gpt c2rcc.msi -h
Prozessierung mit dem C2RCC Prozessor
Stellen Sie eine Terminal-Verbindung mit der VM über ssh her.
Connecting to VM
ssh -i /path/to/your/key.rsa -p 22 eouser@<VM's floating IP>
Aktivieren Sie die virtuelle Umgebung mit der entsprechenden Python-Version.
Activate Environment
conda activate py35
Wir werden unser Skript mit nano erstellen:
Nano Installation
sudo apt install nano
Erstellen wir nun eine ausführbare Python-Datei:
Create Python Script
nano skrypt.py
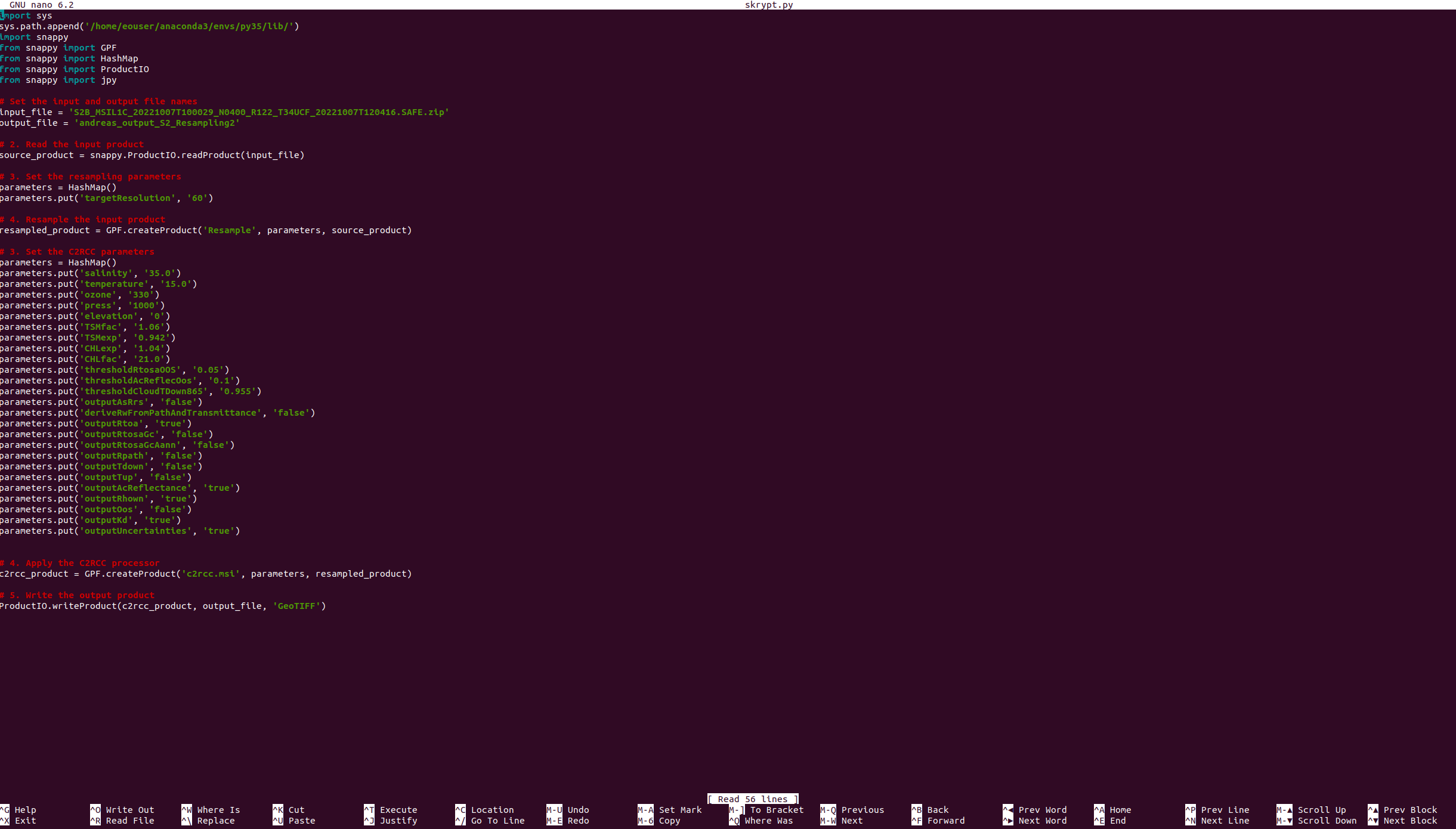
Als Eingabe in das Python-Script wird in unserem Beispiel ein vorbereiteter C2RCC-Code verwendet:
C2RCC python code
import sys
sys.path.append('/home/eouser/anaconda3/envs/py35/lib/')
import snappy
from snappy import GPF
from snappy import HashMap
from snappy import ProductIO
from snappy import jpy
# Set the input and output file names
input_file = '/codede/Sentinel-2/MSI/L1C/2023/04/29/S2A_MSIL1C_20230429T102601_N0509_R108_T32UME_20230429T141009.SAFE'
output_file = 'output_10'
# 2. Read the input product
source_product = snappy.ProductIO.readProduct(input_file)
# 3. Set the resampling parameters
parameters = HashMap()
parameters.put('targetResolution', '10')
# 4. Resample the input product
resampled_product = GPF.createProduct('Resample', parameters, source_product)
# 3. Set the C2RCC parameters
parameters = HashMap()
parameters.put('salinity', '35.0')
parameters.put('temperature', '15.0')
parameters.put('ozone', '330')
parameters.put('press', '1000')
parameters.put('elevation', '0')
parameters.put('TSMfac', '1.06')
parameters.put('TSMexp', '0.942')
parameters.put('CHLexp', '1.04')
parameters.put('CHLfac', '21.0')
parameters.put('thresholdRtosaOOS', '0.05')
parameters.put('thresholdAcReflecOos', '0.1')
parameters.put('thresholdCloudTDown865', '0.955')
parameters.put('outputAsRrs', 'false')
parameters.put('deriveRwFromPathAndTransmittance', 'false')
parameters.put('outputRtoa', 'true')
parameters.put('outputRtosaGc', 'false')
parameters.put('outputRtosaGcAann', 'false')
parameters.put('outputRpath', 'false')
parameters.put('outputTdown', 'false')
parameters.put('outputTup', 'false')
parameters.put('outputAcReflectance', 'true')
parameters.put('outputRhown', 'true')
parameters.put('outputOos', 'false')
parameters.put('outputKd', 'true')
parameters.put('outputUncertainties', 'true')
# 4. Apply the C2RCC processor
c2rcc_product = GPF.createProduct('c2rcc.msi', parameters, resampled_product)
# 5. Write the output product
ProductIO.writeProduct(c2rcc_product, output_file, 'BEAM-DIMAP')
Der obige Code importiert die erforderlichen Module aus der SNAP-Python-API, legt die Namen der Eingabe- und Ausgabedateien fest, liest das Eingabeprodukt aus dem angegebenen eodata-Sentinel-2-Produktpfad, wandelt es auf eine Zielauflösung von 10 Metern um (je höher die Auflösung, desto länger die Dauer der Skriptausführung) und wendet den C2RCC-Prozessor an, um Wasserqualitätsparameter abzuleiten.
Zunächst werden der Pfad der Eingabedatei und der Name der Ausgabedatei festgelegt. Dann wird die Methode snappy.ProductIO.readProduct() verwendet, um das Eingabeprodukt in die Variable source_product zu lesen. Anschließend werden die Parameter für das Resampling, einschließlich der Zielauflösung, mithilfe einer HashMap festgelegt. Dieser Schritt ist notwendig, da für C2RCC alle Spektralbänder eine einheitliche geometrische Auflösung haben müssen. Die neue, einheitliche Rasterung wird durch den Aufruf der Methode GPF.createProduct() mit dem Operatornamen „Resample“, den Parametern und der Variable source_product als Eingaben durchgeführt. Das resultierende, neu gerasterte Produkt wird in der Variablen resampled_product gespeichert.
Anschließend wird die HashMap gelöscht und es werden neue Parameter für den C2RCC-Prozessor festgelegt, einschließlich Wassereigenschaften wie Salzgehalt, Temperatur und Ozon sowie Verarbeitungskennzeichen zur Steuerung der Ausgabetypen. Die Methode GPF.createProduct() wird erneut mit dem Operatornamen „c2rcc.msi“, den Parametern und dem resampled_product als Eingaben aufgerufen, um den C2RCC-Prozessor anzuwenden. Das resultierende Produkt wird in der Variablen c2rcc_product gespeichert.
Schließlich wird die Methode ProductIO.writeProduct() verwendet, um das c2rcc_product in eine Ausgabedatei im BEAM-DIMAP-Format zu schreiben, wobei der Name output_file verwendet wird. Wir empfehlen die Verwendung dieses Formats anstelle von GTIFF, da die virtuellen Bänder mit Nullen gefüllt werden.
Für Ihre eigenen Anwendungen passen Sie den Code entsprechend Ihrer Anforderungen und Ihrer Arbeitsumgebung an. Bitte beachten Sie, dass Nutzende auf dem Daten-Repository /codede nur Leseberechtigung haben.
Fügen Sie den Code ein. Zum speichern der Datei, drücken Sie nacheinander ctrl+X, Y und Enter:
Führen Sie es dann über „py35“ Python aus. Die Prozessierung wird gestartet
Warnung
Bitte beachten Sie vor der Ausführung des Python-Skripts (skrypt.py), dass die Verarbeitungszeit erheblich sein kann und möglicherweise mehrere Stunden in Anspruch nehmen kann.
Führen Sie ein Python-Skript aus
python3 skrypt.py
Herunterladen der Ergebnisse auf Ihre lokale Arbeitsumgebung
Da als Ausgabeformat für die Verarbeitung BEAM-DIMAP gewählt wurde, ist die Beschaffung dieses Formats etwas aufwändiger als im Falle des GeoTIFF-Formats. Das BEAM-DIMAP-Format basiert auf dem Hierarchical Data Format (HDF) und ist für die Speicherung mehrdimensionaler Arrays wissenschaftlicher Daten zusammen mit relevanten Metadaten konzipiert. Es enthält eine Header-Datei mit Metadaten, wie z. B. Geolokalisierungsinformationen, Geräteeigenschaften und Erfassungsparameter, sowie eine Datendatei mit den eigentlichen Bilddaten. Da es sich um ein Mehrdateienformat handelt, müssen wir es herunterladen:
.dim Datei
.data Ordner
Der Ordner .data sollte vor dem Herunterladen komprimiert werden.
Compression of Directory
sudo apt install zip # install zip if necessary
zip -r example.zip <directory name>.data
Laden Sie Dateien auf ihre lokale Arbeitsumgebung, indem Sie die folgenden Befehle lokal ausführen:
Transfer Data
scp -i /path/to/your/key.rsa eouser@<VM's floating IP>:/path/to/dim/file.dim /destination/folder/path/
scp -i /path/to/your/key.rsa eouser@<VM's floating IP>:/path/to/compressed/data/directory/example.zip /destination/folder/path/
Dekomprimieren Sie die Datei example.zip in das Verzeichnis, in dem die .dim-Datei gespeichert ist. Das war’s, die Ausgabe von C2RCC ist bereit für die Verwendung auf Ihrem lokalen Rechner: